세균의 병원성 발현 매커니즘 규명이나 바이오파운드리 기술에서는 DNA에 달라붙는 단백질의 결합 지점을 찾아내 분석하는 기술이 중요한데, 기존보다 매우 적은 시료를 쓰고도 결합 지점을 정확하게 분리해 낼 수 있는 기술이 개발됐다.
울산과학기술원(UNIST) 에너지화학공학과 김동혁 교수팀과 고려대학교 생명과학부 이은진 교수팀은 기존보다 세포를 5000배 더 적게 쓰고도 DNA에서 특수 단백질의 결합 지점을 고해상도로 분석할 수 있는 ‘미니’ 염색질 면역 침강법(ChIP-mini)을 개발했다고 4일 밝혔다.
유전 정보가 저장된 DNA는 염기 분자가 길게 이어진 형태다. 세포는 전사인자라는 특수 단백질을 DNA의 특정 지점에 결합시키는 방식으로 유전자의 발현을 조절한다.
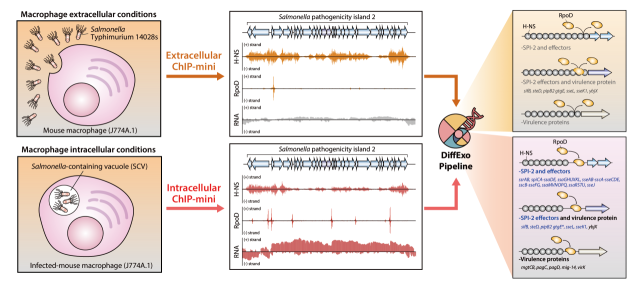
염색질 면역 침강법(ChIP)은 DNA와 결합한 이 특수 단백질만을 분리해 결합된 DNA 조각을 찾는 기술이다. 이번에 개발된 ‘ChIP mini’ 기술은 기존 기술 대비 약 5000배 적은 480만 개의 세포를 이용해서 염기 하나(1 base pair, 약 0.34나노미터) 수준의 정밀도로 결합 지점을 분리해 낼 수 있다. 이 덕분에 근접한 위치에 여러 개의 단백질이 결합해도 각각을 결합 지점을 정확하게 구분해 분석할 수 있다. 기존의 최신 실험인 ChIP-exo 실험법으로는 비슷한 수준의 정밀도를 얻기 위해서 약 100억~1000억 개의 세포가 필요했다.
공동연구팀은 숙주에 감염을 일으킨 극소량의 살모넬라균을 분리해 균 내부에서 일어나는 두 가지 특수 단백질 (H-NS, RpoD)의 DNA 결합 위치와 강도 변화를 정량적으로 분석해 냄으로써 ChIP-mini의 성능을 입증했다. 살모넬라균은 숙주인 대식세포 밖에서는 H-NS 단백질을 DNA에 강하게 결합시켜 병원성 유전자 발현을 억제하지만, 일단 숙주세포로 들어가는 데 성공하면 H-NS 단백질 결합 강도는 줄이고, RpoD 단백질을 결합시켜 병원성 유전자를 선택적으로 활성화한다. 이는 숙주세포 밖에서 불필요하게 병원성을 드러내 숙주의 면역 공격을 받지 않도록 조절하는 방식이다.
살모넬라균은 극미량으로 숙주세포 내부로 감염하기 때문에, 기존 ChIP 실험법들로는 숙주 세포안에 있는 살모넬라균 수가 충분치 못해 이 같은 분석이 힘들었다. 정량적 분석에는 연구팀이 자체 개발한 통계 프로그램인 DiffExo를 썼다.
또 ChIP-mimi 기술의 개별 분석단가는 기존보다 12배 이상 낮춘 2만 원 수준이다.
공동 제1 저자인 박준영 UNIST 박사는 “개발된 기술을 NGS 자동화 플랫폼과 결합하면 바이오파운드리 개발에 필요한 대용량 결합 데이터를 싸고 빠르게 생산하는 우위를 점할 수 있을 것”이라며 “현재 차세대염기서열 분석기(NGS) 자동화 기기와의 호환성 확보를 위한 추가 연구를 수행 중”이라고 설명했다.
바이오파운드리는 세균과 같은 미생물 등을 반도체의 파운드리처럼 돌려서 고부가가치 단백질 등을 생산하는 기술로 미생물 유전자 편집, 최적의 회로설계를 위해서는 대량의 결합 데이터가 필요하다.
김동혁 교수는 “감염성 미생물의 유전자 발현 네트워크 규명과 바이오 부품 발굴 등 바이오파운드리 분야 원천기술로서 가치 있는 연구”라고 설명했다.
이번 연구는 국제학술지 ‘핵산 연구(Nucleic Acids Research)’에 2월 10일 자로 출판됐다. 연구 수행은 과학기술정보통신부의 바이오·의료기술개발과 국가연구재단의 기초 연구실 지원사업의 지원으로 이뤄졌다.
< 저작권자 ⓒ 서울경제, 무단 전재 및 재배포 금지 >








 jjs@sedaily.com
jjs@sedaily.com